Leucémies virales, un nouvel outil prédictif
Communiqué de presse (05/09/2017)
Un nouvel outil prédictif de réponse au traitement pour les leucémies virales
Bruxelles/Liège, le 5 septembre 2017 – Quelques mois après la publication d’un mécanisme inédit par lequel certains virus induisent des leucémies (Nature Communications, 23 mai 2017), l’équipe de recherche dirigée par Anne Van den Broeke (Institut Jules Bordet –Université Libre de Bruxelles et GIGA - Université de Liège) publie une nouvelle étude purement appliquée cette fois. Cette étude pilote, publiée le 5 septembre dans la revue Leukemia décrit une méthode moléculaire permettant de mieux évaluer la réponse au traitement chez des patients atteints d’un type de leucémie particulièrement agressif et en démontre l’utilité pour le suivi clinique. Un nouvel outil qui aidera les cliniciens dans leurs choix de traitements !
De la recherche fondamentale à la recherche appliquée
Dans l’étude fondamentale qui a mené à la découverte d’un nouveau mécanisme par lequel certains virus induisent des leucémies (publiée en mai 2017), l’équipe d’Anne Van den Broeke s’est intéressée à la leucémie induite par le virus HTLV-1 (Human T-cell leukemia virus) ainsi qu’à un modèle animal correspondant associé à l’infection par BLV (le virus de la leucémie bovine, qui provoque des leucémies chez le bovin et le mouton). Dans le cadre de ces travaux, les chercheurs ont développé des méthodes basées sur des technologies de séquençage à haut débit, dans le but de mieux comprendre le rôle du virus dans la progression tumorale. Ils se sont rapidement aperçus que l’une d’entre elles, basée sur l’identification de la position du virus dans le génome (matériel génétique) de son hôte, pouvait être utile dans le suivi thérapeutique des patients atteints de leucémie causée par HTLV-1. Ils se sont alors attelés à l’optimiser.
Une méthode applicable en clinique
Chaque cas de leucémie est caractérisé par une position bien précise du virus dans le génome des cellules du sang, différente pour chaque patient. La méthode permet de détecter cette « carte d’identité virale » de manière quantitative, sa diminution étant le reflet d’une bonne réponse au traitement, son augmentation signant une recrudescence de la leucémie et donc la rechute pour le patient. Grâce aux améliorations apportées à la technique initiale, les chercheurs sont parvenus à obtenir un outil moléculaire nettement plus performant et plus sensible.
D’autre part, ils ont réussi à en réduire les coûts au point où il est envisageable de l’utiliser en clinique. L’étude pilote publiée dans Leukemia décrit le suivi de patients par cette méthode moléculaire optimisée. Les résultats montrent qu’elle permet, pour la première fois, de mieux évaluer la réponse au traitement, de prédire une rechute précoce et par conséquent, d’aider les cliniciens dans leurs décisions thérapeutiques. Jusqu’à présent, ces patients, pour lesquels le pronostic est malheureusement très défavorable, ne bénéficiaient d’aucune méthode fiable pour évaluer la réponse thérapeutique. L’étude montre que la méthode est capable de détecter une rechute là où elle échappait à la détection par des méthodes conventionnelles. Les travaux, réalisés en collaboration avec l’Hôpital universitaire Necker à Paris, sont un bel exemple de recherche fondamentale débouchant sur une application clinique. L’étape suivante sera de définir les conditions optimales d’utilisation de cet outil en routine clinique afin de l’intégrer au mieux au bénéfice du patient.
- Références de l’étude:
Maria Artesi, Ambroise Marçais, Keith Durkin, Nicolas Rosewick, Vincent Hahaut, Philippe Suarez, Amélie Trinquand, Ludovic Lhermitte, Véronique Avettand Fenoel, Michel Georges, Olivier Hermine & Anne Van den Broeke
Monitoring molecular response in Adult T-cell Leukemia/Lymphoma by high throughput sequencing analysis of HTLV-1 clonality, Leukemia, 2017, doi:10.1038/leu.2017.260
- Chercheurs senior :
Dr Anne Van den Broeke (Institut Jules Bordet et GIGA-R) et Prof Michel Georges (GIGA-R). Laboratoire d’Hématologie expérimentale, Institut Jules Bordet, Université Libre de Bruxelles (ULB), Boulevard de Waterloo 121, 1000 Brussels, Belgium.
Unité de Génomique animale, GIGA-R, Université de Liège, Avenue de l’Hopital 11, B34, 4000 Liège, Belgium.
- Financement de l’étude :
Ce travail a été financé par les Amis de l’Institut Bordet, le Fonds de la Recherche Scientifique (FRS), Télévie, l’International Brachet Stiftung (IBS) et la Fondation Lambeau Marteaux.
- Collaborations internationales
Hôpital universitaire Necker, Paris, France – Dr Olivier Hermine et Dr Ambroise Marçais
- Pièce jointes :
Illustration du suivi des patients
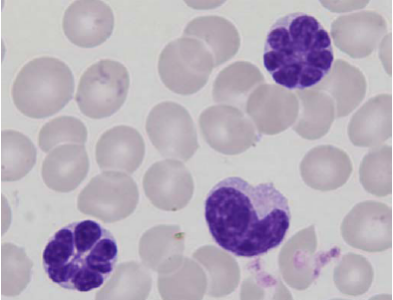
Photo : Globules blancs en forme de fleur caractéristiques de la leucémie virale
- Pour plus d’information:
Dr Anne Van den Broeke: anne.vandenbroeke@bordet.be
Prof Michel Georges: michel.georges@ulg.ac.be
Contacts Presse
- Institut Jules Bordet
Ariane van de Werve
Tel: +32 2 541 31 39
GSM : +32.48617 33 26
E-mail : ariane.vandewerve@bordet.be
www.bordet.be
- Université de Liège
Didier Moreau
Tel +32 4 366 52 17
GSM : +32 494 572 530
E-mail : dmoreau@ulg.ac.be
A télécharger
- Institut_Jules_Bordet_Leucemie_nouvel_outil_prédictif_reponse_traitement.pdf136.84 Ko
- Institut_Jules_Bordet_Novel_tool_monitor_therapeutic_response_patients_viral_leukemia.pdf145.53 Ko
- Jules_Bordet_Instituut_nieuwe_tool_voorspellen_behandelingsrespons_virale_leukemietypes.pdf147.05 Ko
- Flower_cells_ATL.jpg106.31 Ko
- Leucemie_Illustration_suivi_patients.pdf260.16 Ko
- Leukemia_Patients_follow-up_illustration.pdf338.99 Ko